library("tidyverse")
library("cowplot")
library("lubridate")
library("viridis")
path <- url("https://raw.githubusercontent.com/mebucca/dar_soc4001/master/slides/class_12/covid_data.csv")
# leer archivo csv
covid_data <- read_delim(path, delim=";")
covid_data <- covid_data %>% select(continent, total_cases_per_million, total_deaths_per_million, new_cases_per_million, new_deaths_per_million)SOC4001 Procesamiento avanzado de bases de datos en R
Ponderación: 12% de la nota final del curso
Formato: Desarrollar esta tarea en un RScript, agregando comentarios cuando sea necesario.
Instrucciones:
Usa el siguiente cógigo para cargar la base de datos sobre Covid-19 usados en clase y seleccionar sólo las variables numéricas más el identificador de continente.
Referencia: Hasell, J., Mathieu, E., Beltekian, D. et al. A cross-country database of COVID-19 testing. Sci Data 7, 345 (2020). https://doi.org/10.1038/s41597-020-00688-8 y utilizad
Los datos deben verse así:
Rows: 56,748
Columns: 5
$ continent <chr> "Asia", "Asia", "Asia", "Asia", "Asia", "Asia…
$ total_cases_per_million <dbl> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, N…
$ total_deaths_per_million <dbl> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, N…
$ new_cases_per_million <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
$ new_deaths_per_million <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …Ejercicio
- Usando las funciones
nest()ymap()del paquetepurrr, para cada continente crea un matriz de correlaciones entre todas las variables.
- Nota: para crear la matriz de correlaciones la función
map()debe tomar el siguente argumento:.f = ~ cor(., use = "pairwise.complete.obs"). Guarda la matriz en una nueva columna llamadacorrsy asigna el resultado a un nuevo objecto llamadomiscors.
El objeto miscors debe verse así:
# A tibble: 6 × 3
continent data corrs
<chr> <list> <list>
1 Asia <tibble [13,528 × 4]> <dbl [4 × 4]>
2 Europe <tibble [14,828 × 4]> <dbl [4 × 4]>
3 Africa <tibble [13,637 × 4]> <dbl [4 × 4]>
4 North America <tibble [9,116 × 4]> <dbl [4 × 4]>
5 South America <tibble [3,404 × 4]> <dbl [4 × 4]>
6 Oceania <tibble [2,235 × 4]> <dbl [4 × 4]>y cada matriz de correlación contenida en corrs de ve así
total_cases_per_million total_deaths_per_million
total_cases_per_million 1.00000000 0.02057843
total_deaths_per_million 0.02057843 1.00000000
new_cases_per_million 0.16873694 0.06326542
new_deaths_per_million 0.16440194 -0.13163684
new_cases_per_million new_deaths_per_million
total_cases_per_million 0.16873694 0.16440194
total_deaths_per_million 0.06326542 -0.13163684
new_cases_per_million 1.00000000 -0.02377809
new_deaths_per_million -0.02377809 1.00000000- Continua trabajando con el objeto
miscors. Usando las funcionesnest()ymap()del paquetepurrr, crea nueva columna llamadamean_corque contenga el promedio de cada matriz de correlacciones almacenada en la columnacorrs.
- Nota: para el promedio de las correlaciones la función
map()debe tomar el siguente argumento:.f = ~mean(., na.rm=T). Asigna el resultado a un nuevo objecto llamadomiscors. El nuevo objetomiscorsdebe verse así:
# A tibble: 6 × 4
continent data corrs mean_cor
<chr> <list> <list> <list>
1 Asia <tibble [13,528 × 4]> <dbl [4 × 4]> <dbl [1]>
2 Europe <tibble [14,828 × 4]> <dbl [4 × 4]> <dbl [1]>
3 Africa <tibble [13,637 × 4]> <dbl [4 × 4]> <dbl [1]>
4 North America <tibble [9,116 × 4]> <dbl [4 × 4]> <dbl [1]>
5 South America <tibble [3,404 × 4]> <dbl [4 × 4]> <dbl [1]>
6 Oceania <tibble [2,235 × 4]> <dbl [4 × 4]> <dbl [1]>- Usando la función
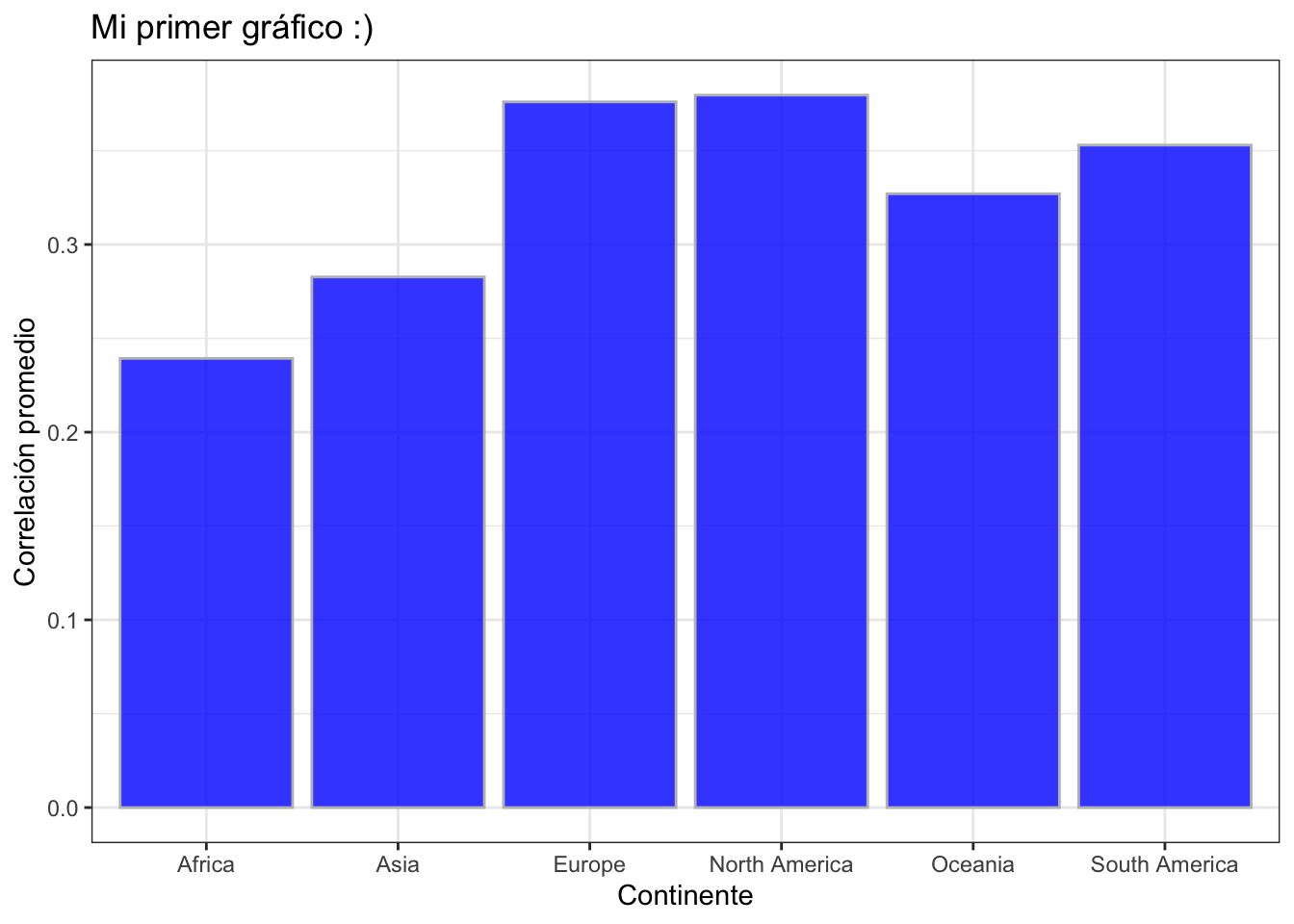
unnest()crea un nueva base de datos que contenga solo la correlación promediomean_corpor continente. Asigna el resultado a un nuevo objecto llamadomiscors. El nuevo objetomiscorsdebe verse así:
# A tibble: 6 × 2
continent mean_cor
<chr> <dbl>
1 Asia 0.283
2 Europe 0.376
3 Africa 0.239
4 North America 0.380
5 South America 0.353
6 Oceania 0.327- En base a los datos almacenados en
miscorscrea el siguiente gráfico usando la geometríageom_bar(stat = "identity"):